오늘은 AWS HealthOmics 서비스에 대해 알아보겠습니다.
AWS HealthOmics는 연구자들이 genomics와 같은 다양한 바이오 데이터를 분석하는데 도움이 되는 AWS 서비스입니다.
HealthOmics에는 다음과 같이 크게 3가지 요소가 있습니다.
- 📦 HealthOmics Storage : Genomic data를 저장 및 공유할 수 있는 storage (Petabyte 규모)
- 📝 HealthOmics Analytics : Genomic variants 및 annotation 데이터를 변환 & 분석
- 🧬 HealthOmics Workflows : Workflow를 사용하여 genomic data를 분석
(📝 HealthOmics Analytics 의 경우, 분석에 대한 내용이기에 다음 포스팅에서 다루도록 하겠습니다.)
그럼 각 요소에 대해 하나씩 살펴보겠습니다.
================================================================================
🔍 구성요소
================================================================================
📦 HealthOmics Storage
HealthOmics Storage는 genomic data를 효율적이고 저렴하게 저장, 검색, 구성, 공유를 할 수 있게 해줍니다.
이 때, HealthOmics Storage의 경우 다음과 같은 특성이 있습니다.
- 30일동안 액세스하지 않은 ACTIVE 데이터는 자동으로 ARCHIVE 상태에 저장
- Read-set(읽기 세트) 객체로 저장
- Read-set : Sequencing 파일 뿐만 아니라 메타데이터도 함께 저장되어 있는 객체
🔖 ETag (Entity Tag)
- Sequence Store에 저장된 파일의 무결성을 확인/추적하기 위한 해시값
- 파일 내용을 기반으로 생성되기에 메타데이터 변경에는 영향을 받지 X
- Sequence Store에서는 파일이 압축/해제되어도 Etag는 동일
- 알고리즘 : MD5(기본), SHA-256, SHA-512
⚠️ Amazon S3 ETag와는 다른 점을 유의하세요!
- Amazon S3 ETag : Bit 단위의 동일성 (bitwise identity)
- HealthOmics ETag : 파일의 의미적 동일성 (압축 여부 상관 없이 동일)
📦 Reference Store
- Genome reference 저장 (ex. hg19.fa)
- 💰 무료
📦 Sequence Store
- Raw sequencing data 저장 (ex. NA12878.fastq.gz)
- 💰 Gigabase 당 과금
================================================================================
🧬 HealthOmics Workflows
HealthOmics에서 제공하는 workflow는 다음과 같이 크게 2가지 종류가 있습니다.
—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–
🔍 Ready2Run
Ready2Run은 Third-party publisher가 미리 구성해놓은 workflow를 사용할 수 있는 서비스입니다.
GATK-BP부터 AlphaFold까지 다양한 workflow가 미리 만들어져있어서 손쉽게 분석을 해볼 수 있습니다.
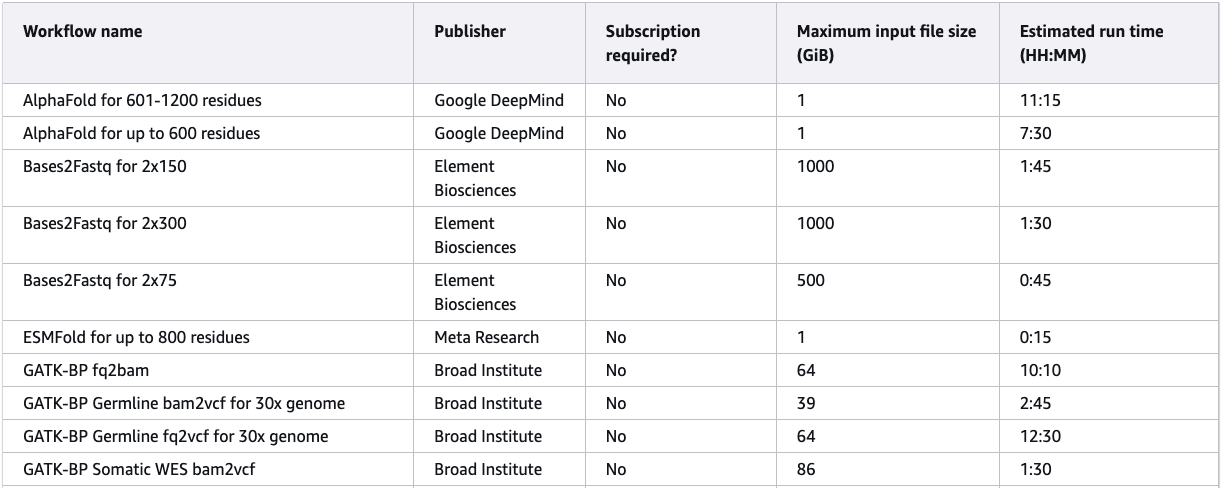
일부 workflow(Sentieon)의 경우에는 별도의 구독이 필요하기도 합니다.
자세한 내용은 🔍 Ready2Run Workflow 부분을 참고하세요.
🔒 Private Workflow
Private workflow는 사용자가 자신의 개인적인 workflow를 실행하고자 할 때 사용할 수 있는 서비스입니다.
이 때, 지원되는 workflow 언어는 ① WDL ② CWL ③ Nextflow 이렇게 3가지가 있습니다.
—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–
🧐 Run Analyzer
Private Workflow를 실행할 때, 최적화를 도와주는 오픈소스 툴입니다.
크게는 다음과 같은 역할을 합니다.
- 메모리 & CPU의 병목현상 평가
- 메모리 & CPU가 over-provision된 부분을 찾아 비용을 절감할 수 있는 새로운 인스턴스 크기 권장
- 작업의 타임라인 뷰를 제공
- 실행 스토리지에 대한 권장사항을 제공
================================================================================
💰 비용
================================================================================
AWS HealthOmics는 AWS Free Tier를 사용하여 무료로 시작해볼 수 있습니다.
처음 2개월간은 다음과 같이 Free Tier를 사용할 수 있습니다.
—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–
🧬 HealthOmics Workflows
- omics.m.xlarge(또는 동급) 인스턴스 최대 275시간 및 49,000GB의 실행 스토리지
- omics.m.xlarge : vCPU 4 & 메모리 16GiB
📦 HealthOmics DataStore
- Sequence Store
- Active storage/archive storage모두 1,500 gigabase/month
- Variant Store
- 200 GB/month
⚠️ Sequence Store는 Gigabase/Variant Store는 GB(Gigabyte) 기준으로 과금되는 점을 참고하세요!
—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–
이후에는 사용자의 사용량에 따라 과금💰이 됩니다.
1. Private Workflow 실행
- Workflow Task Instance
- 각 Task에 요청된 사양(ex. vCPU, 메모리)를 충족하는 가장 작은 omics instance에 매핑
- Run Storage
- Static : 1,200 GiB → 2,400 GiB→ 이후 2,400 GiB 단위로 지정 가능
- Dynamic : 사용량에 따라 자동 조정
📌 Workflow 실행 중(Running) → 💰 부과 O
📌 Workflow 보류 중(Pending), 시작(Starting), 중지(Stopping) → 💰 부과 X
2. Ready2Run Workflow 실행
- 선택한 Workflow당 요금 부과
📌 실행이 성공적으로 완료되면 실행 시간에 관계없이 실행당 동일한 고정 요금 💰 부과
3. Data Store
- 대규모 샘플 데이터를 위한 관리형 FAIR(검색 가능, 액세스 가능, 상호 운용 가능, 재사용 가능) 스토리지
- 월별 Gigabase당 요금 💰 부과 → 파일 형식/압축에 대해 고민 필요 X
- HealthOmics API를 통해 read-set에 접근할 때, GET requests을 제외한 모든 요청은 무료
4. Data Transfer
—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–o—–
자세한 내용은 💰 AWS HealthOmics Cost 부분을 참고하세요.
================================================================================
👍🏻 장점
================================================================================
그래서 AWS HealthOmics 서비스가 기존의 AWS 서비스들보다 어떤 우수한 부분이 있는지 설명드리겠습니다.
AWS HealthOmics와 비교할만한 서비스로는 AWS Parallel Cluster와 AWS Batch가 있지만, 이들보다 더 유리한 점들이 있습니다.
🧑🏻🔬 연구자들의 간편한 사용 가능
- [Parallel Cluster & Batch] 대량의 분석에 필요한 HPC 구성을 위해 직접 클러스터 구성이 필요
- [HealthOmics] 👍🏻
- 별도의 클러스터 구성 없이 바로 사용할 수 있도록 구성되어있음
- 연구자들이 직접 인프라를 구성하거나 관리할 필요가 없음
🧬 Workflow 최적화
- [Parallel Cluster & Batch] 범용적인 HPC 및 배치 처리 용도로 설계되었기에 최적화 부족
- [HealthOmics] 👍🏻
- NGS 분석, Variant calling 등 Ready2Run workflow를 사용하여 바로 분석할 수 있음
- Private workflow도 개인적인 Workflow가 있을 경우, 손쉽게 분석을 진행할 수 있음
💰 비용 효율적
- [Parallel Cluster & Batch] 불필요한 노드가 유지될 수 있고, 혹은 비용 최적화를 위한 설정이 복잡할 수 있음
- [HealthOmics] 👍🏻
- 분석에 적합한 최소한의 서버를 자동으로 할당해주고, run analyzer를 사용하여 최적화도 가능
- 분석이 완료되면 자동으로 자원을 해제하기에 불필요한 비용 절감 가능
📦 데이터 관리 최적화
- [Parallel Cluster & Batch] S3와 같은 스토리지와 별도의 연결 필요
- [HealthOmics] 👍🏻
- Fastq와 같이 bioinformatic 관련 데이터를 다루기에 편한 스토리지 제공
- 압축의 종류나 여부와 상관없이 같은 파일이라고 인식됨
📃 Reference
- 🔍 Ready2Run Workflow : https://docs.aws.amazon.com/omics/latest/dev/workflows-r2r-table.html
- ✍🏻 AWS HealthOmics HandsOn : https://catalog.workshops.aws/amazon-omics-end-to-end/en-US?refid=ap_card
- 💰 AWS HealthOmics Cost : https://aws.amazon.com/ko/healthomics/pricing/
SA 서희재
![Read more about the article [AWS Summit Korea 2025] 생성형 AI 기반으로 스마트 인사이트 구현](https://tech.cloud.nongshim.co.kr/wp-content/uploads/image-420.png)